近日,江南大学陈兴教授课题组在国际知名期刊Molecular Therapy上发表了题为“DeepHEM: A Novel Deep Domain-Adversarial Learning Framework for Identifying Human Essential MiRNAs”的论文。该研究在人类必需微小核糖核酸(miRNA)识别方向取得重要进展。团队创新性地提出了深度领域对抗学习框架DeepHEM,成功攻克了传统方法中存在的实验标签缺失、跨物种知识迁移受限以及多模态特征融合不充分等关键难题,为基于RNA靶点治疗的候选药物分子筛选工作提供了高效且精准的计算工具。此期刊在中科院期刊分区中大小类均为一区Top,最新影响因子为12。陈兴教授为该论文的通讯作者。
miRNA作为调控基因表达的关键非编码RNA分子,在细胞增殖、胚胎发育、造血分化等基础生理过程中发挥着不可替代的作用,同时也是RNA靶点治疗的核心靶点。它既能作为疾病早期诊断的生物标志物,也是开发精准疗法的“分子开关”。然而,传统通过基因敲除实验来识别必需miRNA的方法,受到高成本、长周期以及伦理约束等因素的限制,难以在人类中开展大规模筛选工作。与此同时,由于缺乏人类必需miRNA的实验验证标签,人类必需miRNA的精准识别长期面临瓶颈,这严重制约了RNA靶向治疗的研发进程。针对这一难题,陈兴教授团队创新性地提出了DeepHEM框架,构建了“多模态特征提取 + 多损失优化”的双核心设计。该设计成功将必需miRNA的标签知识从小鼠(源域)迁移至人类(目标域),进而实现了跨物种知识的高效迁移以及人类必需miRNA的精准预测。实验结果显示,DeepHEM的预测性能远超现有主流方法,充分证明了其架构设计的合理性与可靠性。生物相关性分析表明,具有较高必需性得分的miRNA不仅与更多疾病相关联,还具有更强的进化保守性。在对比实验中,DeepHEM在疾病谱宽度分数和保守性分数这两项生物指标的相关性分析中,显著优于传统域适应方法、深度域混淆方法以及深度适应网络,其预测结果中有80%可通过现有实验文献进行验证。更为重要的是,DeepHEM为RNA疗法研发提供了“低成本预筛选工具”。传统方法需要耗费数月至数年开展敲除实验来验证候选的必需miRNA,而DeepHEM能够快速预测人类miRNA的必需性,优先筛选出高潜力靶点,大幅缩短了湿实验周期。同时,其框架还可扩展至长链非编码RNA、环状RNA等其他非编码RNA的必需性预测,为泛RNA疗法研究奠定了基础。
该研究工作获得了国家自然科学基金重大研究计划培育项目(项目号:92370131)和无锡市太湖人才计划创新领军人才项目(A类项目)的资助。近年来,陈兴教授团队致力于“人工智能 + 药物研发”交叉领域的研究,在非编码RNA药物靶点预测、药物-药物相互作用预测、药物-靶点亲和度预测、增效组合药物预测等前沿方向开展了系统性研究,取得了丰硕的成果,相关研究发表于Molecular Therapy、Briefings in Bioinformatics、IEEE Journal of Biomedical and Health Informatics、Nucleic Acids Research等本领域权威期刊。
论文链接:https://www.sciencedirect.com/science/article/abs/pii/S152500162500958X
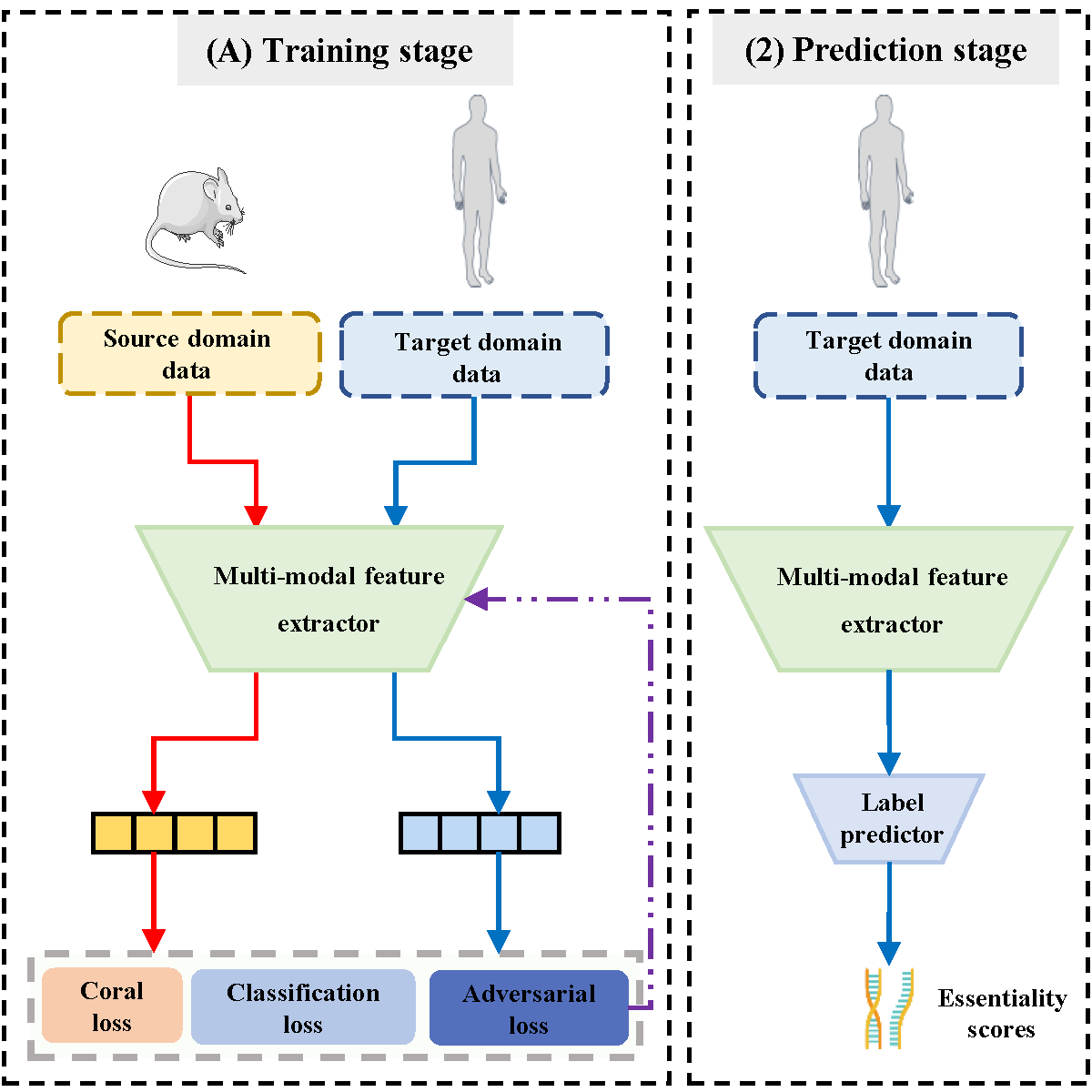
图1 摘要图
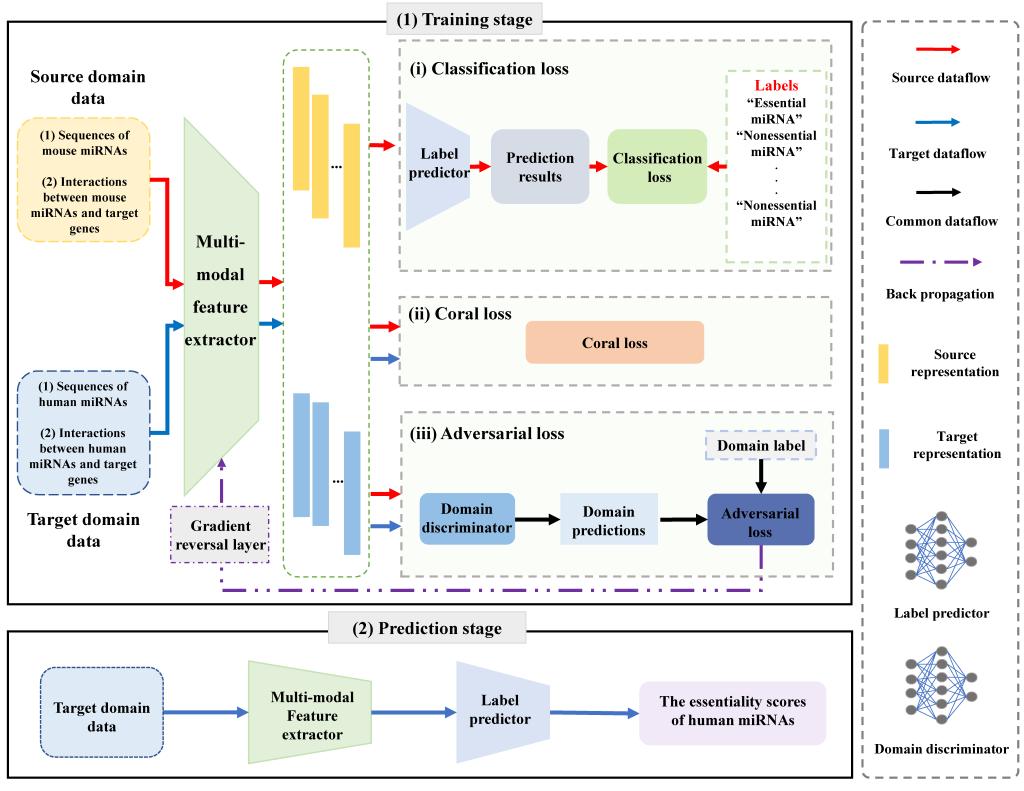
图2基于深度领域对抗学习框架的人类必需miRNA识别方法流程图
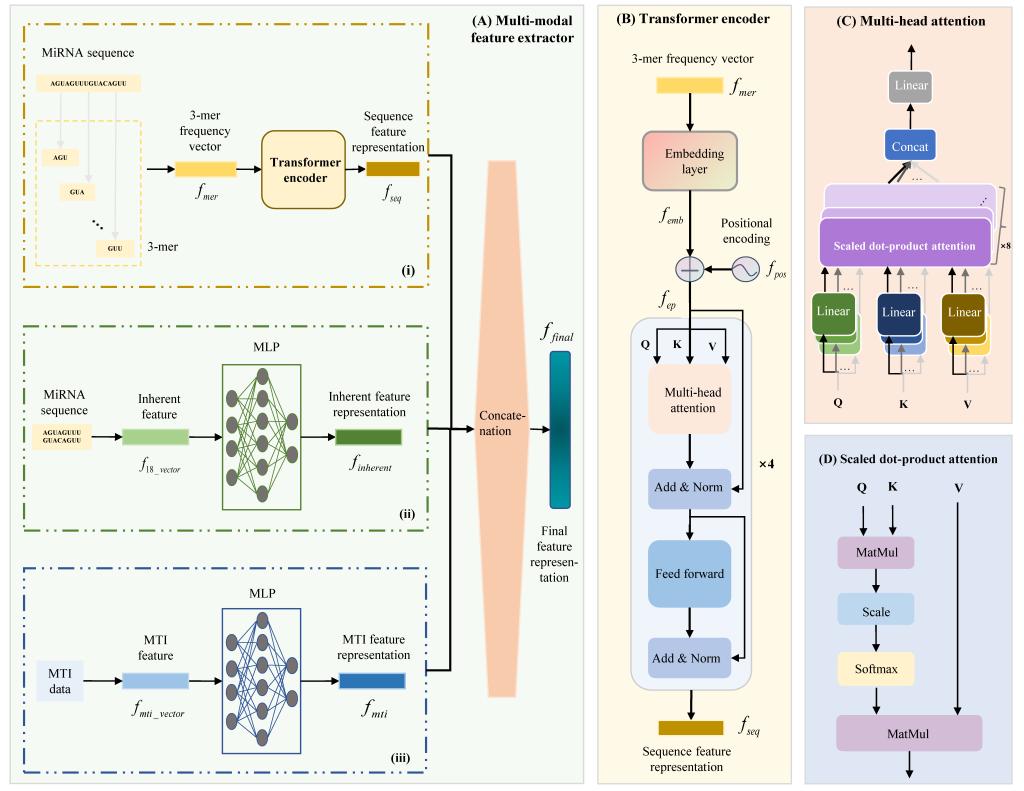
图3 多模态特征提取器结构











